AlphaFold2是deepmind发表在nature上的一篇文章。据说,发表周期非常快(加急预览:Nature的Accelerated Article Preview),可能是因为是同一天,science发表了另一外来自华盛顿大学的另一个竞争工作(RoseTTAFold)。nature抓紧把deepmind这篇也快速见刊,使得更有噱头(猜测)。
RoseTTAFold文章见:https://science.sciencemag.org/content/early/2021/07/14/science.abj8754;
RoseTTAFold代码见:https://github.com/RosettaCommons/RoseTTAFold
AlphaFold2的论文见:https://www.nature.com/articles/s41586-021-03819-2_reference.pdf
代码见:https://github.com/deepmind/alphafold
蛋白质对生命很重要,知道它们的结构可以促进人们对蛋白质的功能有更系统的理解。目前来说,人们已经知道10万种蛋白质的结构。(Proteins are essential to life, and understanding their structure can facilitate a mechanistic understanding of their function. Through an enormous experimental effort, the structures of around 100,000 unique proteins have been determined)
但是人们已经知道的蛋白质序列有按以十亿计。人们只知道它们的系列,而不知道它们的结构。(but this represents a small fraction of the billions of known protein sequences)
给定蛋白质的核酸序列,预测其3D结构是一个重要开放性问题,已经被研究超过半个世纪了。(Predicting the 3-D structure that a protein will adopt based solely on its amino acid sequence, the structure prediction component of the ‘protein folding problem’8 , has been an important open research problem for more than 50 years)
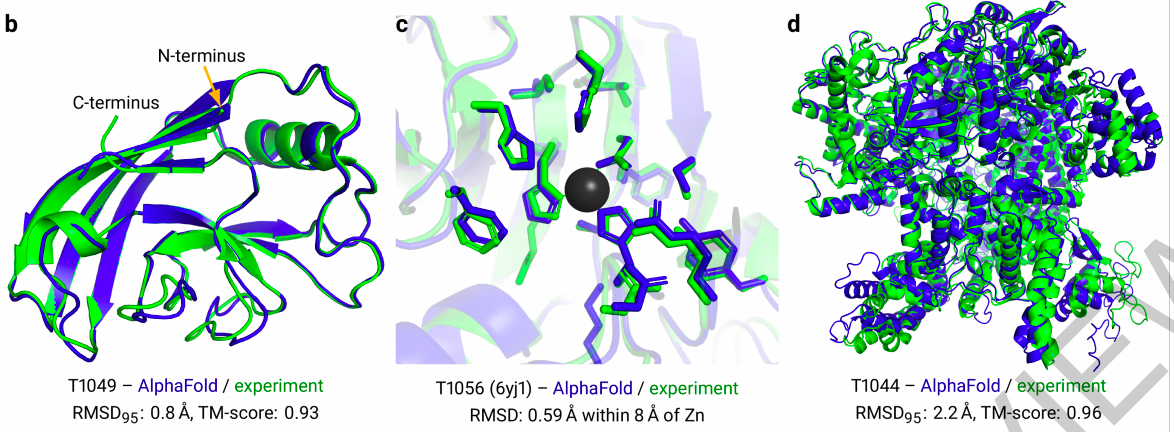
首先来看一下效果:在下图中,蓝色是AI预测结果,绿色是人类实验结果。可以看出来,两者高度接近。尤其是d图中,这么长的序列都做到这种效果,让人佩服。
接下来介绍一下它的主要方法。它的方法其实很简单,如下图所求:
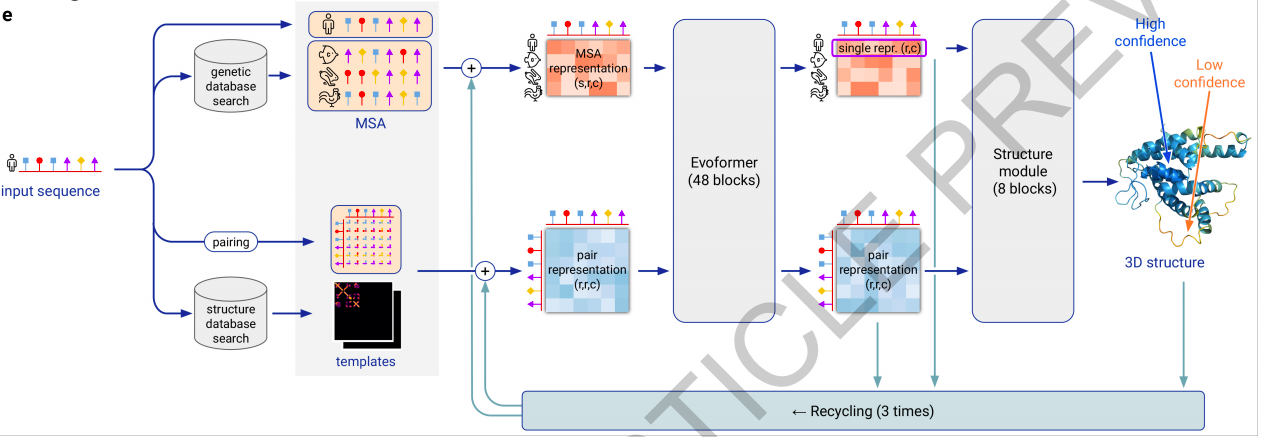
上图看起来挺复杂,其实很简单。首先,输入是一个蛋白质序列(也许或者专业称为蛋白质的核酸序列,即图中的人序列)。然后,这个序列输到两个分支。
在上面这个分支中,我们用这个蛋白质的核酸序列去一个数据库中进行搜索,得到与它相关(例如是相似?)的若干条系列(图中有3条,分别为鱼序列、兔序列和鸡序列)。再加上输入的人序列,共计有四个序列。文章中记序列长为N_seq (图中N_seq = 6),记序列个数为N_res (图中N_res = 4)。然后,这4条长度为6的序列(多次循环中称为 multiple sequence alignments (MSAs)),输入到一个叫做Evoformer中(Transformer的一种),最终输入同样是4条长度为6的序列。
在下面这个分支中,我们人序列求一个类似协方差矩阵一样的矩阵(图中称为pairing),得到一个6x6的矩阵。同时,也用这个人序列去一个数据库中进行搜索,得到若干个人序列pairing矩阵相关(例如是相似?)的若干个template。然后人的paring矩阵(多次循环中称为pairwise features)与template一块输入到Evoformer中,最终输出同样是6x6的矩阵。
然后,上述两个分支的输出(4条长度为6的序列和一个6x6的矩阵)经过上述过程循环3次,最终它们被输入到一个structure module里面,输入一个旋转角度和一个平移(初始化时,所有的元素(我称为元素)的旋转角度设为相同,所有的平移都设在原点)。即可预测蛋白质的空间结构了。(The trunk of the network is followed by the Structure Module that introduces an explicit 3-D structure in the form of a rotation and translation for each residue of the protein (global rigid body frames))
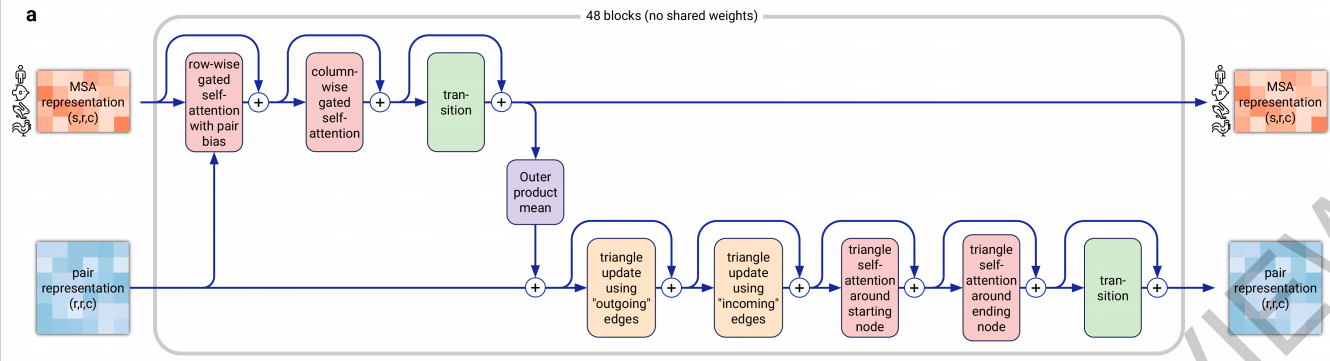
这篇文章的一个贡献是Evoformer,其实非常简单,就是一个Transformer而已。具体网络结构如下图所示,这里就没有必要详细展开介绍了,因为打字很辛苦。
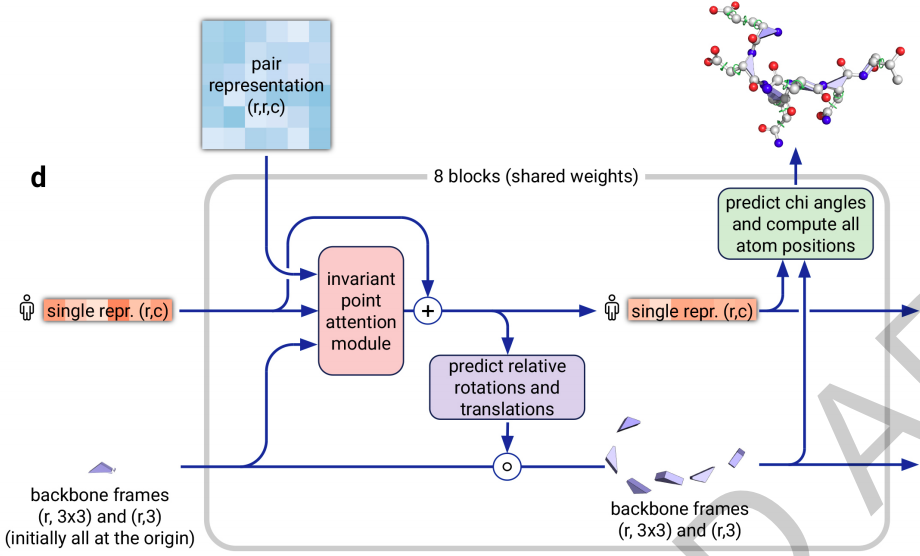
这篇文章的另一个贡献是structure module,其实也非常简单,就是一个加了一个attention模块的普通网络。具体网络结构如下图所示,这里就没有必要详细展开介绍了。显然,旋转分量和平移分量都是N_res个,因为序列的长度就是N_res嘛。(The 3-D backbone structure is represented as N_res independent rotations and translations each with respect to the global frame (“residue gas”, Fig. 3e). )
上面已经基本介绍完网络结构了。接下来介绍如何训练这个网络。训练过程也是有一定的技术,充分地利用了有标签和无标签数据。首先用有标签数据进行了训练一个初步模型(如果大家没有忘记我上面写的,目前来说,人们已经知道10万种蛋白质的结构,所有有标签的数据是很少地,就算把全人类所了解的10万个结构都算上,也才10万。何况,我觉得DeepMind应该也获取不了人类全部知道的10万个数据,因此,有标签的数据量是小于10万的)。然后,作者用这个初步模型来对无标签数据打伪标签,共打了35万个标签。大家看看,这样子数据量一下子就大起来了。难怪DeepMind能在本次蛋白质比赛中取得第一名且遥遥领先。除此之外,作者还作了数据增广(Data augmentation),使用的增广技术包括cropping and MSA subsampling 。
除了上面所提到的自蒸馏技术(即打伪标签技术),作者还使用了最新流行的自监督技术。具体地,在获取上述multiple sequence alignments (MSAs)特征时,使用了BERT来提取特征。具体地,MAS特征,是用BERT(一种Transformer)来提取的。那么,训练这个BERT时,就可以像自然语言中那样,先mask掉一些位置,再预测所mask掉的位置(即masking自监督技术)。
作者接着用一段落Interpreting the neural network来作ablation study和component analysis来解释transformer学习和各种行为。作者也用了一个段落MSA depth and cross-chain contacts来解释了影响精度的因素。
最后两段分别是related work和discussion。其中discussion很长,讨论了本文对现在和未来的重要性。
补充材料主要包括两部分:
第一部分是Method:
包括算法细节(Full algorithm details)
网络细节(Invariant Point Attention (IPA))
输入与数据来源细节(Inputs and data sources (including BFD))
训练细节(Training regimen):128 TPUv3 cores, The initial training stage takes approximately one week, and the fine-tuning stage takes approximately 4 more days.
测试细节:on V100 GPU are 4.8 minutes at 256 residues, 9.2 minutes at 384 residues, and 18 hours at 2,500 residues
评价标准:Metrics
报告总结:Reporting summary:Further information on research design is available in the Nature Research Reporting Summary linked to this paper
第二部分是Data availability
输入数据集在哪下载:All input data are freely available from public sources
第三部分是 Code availability
源代码在哪里下载
软件在哪下载如tensorflow
其他专业软件在哪里下载
数据分析软件如Python3.6在哪里下载
这篇文章的主要代码由tensorflow等写成:
例如Evoformer在这:https://github.com/deepmind/alphafold/blob/d26287ea57e1c5a71372f42bf16f486bb9203068/alphafold/model/modules.py
structure module在这:https://github.com/deepmind/alphafold/blob/main/alphafold/model/folding.py
-----------------------------------
大家好,我来自fast lab。我开始不定时公开写作。这些写作主要通过两个渠道公布:一是FAST LAB官方网站;一是印象识堂(微信可访问)。欢迎大家订阅。谢谢!
FAST Lab的官方网址为:https://wanggrun.github.io/projects/fast
除此外,还可以关注我的小伙伴王广润:https://wanggrun.github.io/
王广聪: https://wanggcong.github.io/
石阳:https://www.linkedin.com/in/%E9%98%B3-%E7%9F%B3-381b521a4/
有时候这些网站打不开,请耐心多点几次。
多谢大家关注。